2026-01-27 17:46:00
对因果推断的常见算法进行总结与实战分析,整体行文重视思路的引导,配合丰富 Python 代码示例,非常适合新手入门和上手;文章后半部分的进阶内容包含作者对因果推断先进方法的看法与总结,虽然在数学理论上不够严密,但作者将个人的推演思路和实践经验进行了很好地展示
内容深入浅出,既适合入门,也值得深挖
全文配套代码,理论结合实践,实用性强
文章思路前后贯通,但知识体系有些凌乱
来上一杯顶着厚泡沫的冰镇啤酒,假设顶上的厚泡沫是数据科学,那啤酒则是统计学基础、科学好奇心和对复杂问题的热情,我们应该学习和关注更实质的存在,而需要忽视泡沫
莫弗(Moivre)方程: $$SE=\frac{\sigma}{\sqrt{ n }}$$
因果图以变量的独立性和条件独立性假设为基础,描述变量间的因果关系
COP 效应 $$ \begin{align*} &E[Y|T=1] - E[Y|T=0] \\ &=E[Y|Y>0, T=1]P(Y>0|T=1) - E[Y|Y>0, T=0]P(Y>0|T=0) \\ &=\underbrace{{P(Y>0|T=1) - P(Y>0|T=0)}}_{Participation \ Effect} * E[Y|Y>0, T=1] \\ &+\underbrace{{E[Y|Y>0, T=1] - E[Y|Y>0, T=0]}}_{COP \ Effect} * P(Y>0|T=0) \end{align*} $$
控制选择偏差: (1)避免引入干预与结果路径之间的中介特征 (2)避免引入同时受到干预和结果影响的特征
非服从性:受试者可能不服从安排和干预,比如医疗实验中的部分倔小孩
内部效度:在特定场景下可识别到的有效因果,比如工具变量法给出的 ATE
外部效应:将当前样本中发现的效应推广到其他人群中,关注效应的预测能力
局部平均干预效应(LATE)
工具变量法的结果只是对具有服从行为对象的局部平均干预效应(LATE)
子分类估计器(Subclassification Estimator)
$$ATE=\Sigma_{i=0}^K(Y_{k_{1}}-Y_{k_{0}}) \times N_{k}/N$$
由于维度诅咒的存在,子分类估计器在实践中应用较少
匹配估计器:为每个干预组样本找到最相似的样本,构建对照组
机器学习
将业务问题转化为预测问题,那么机器学习就会是合适的工具
干预效应不仅与结果变量相关,还与干预水平相关
导致干预效应异质性的关键复杂因素是结果函数 $Y(0)$ 的非线性
合成双重差分法 SDID:融合双重差分法 DID 和合成控制法的优势
英文原本(全)《Causal Inference for The Brave and True》 中文译本(仅前 21 章,且更新不及时)《因果推断:从概念到实践》
2026-01-21 18:59:00
前置知识:双重差分法 DID、合成控制法、双向固定效应 TWFE
已知双重差分法 DID 的线性模型可表示如下: $$ Y_{it} = \beta_0 + \beta_1 Post_t + \beta_2 Treated_i + \beta_3 Treated_i Post_t + e_{it} $$
post 表示时间虚拟变量,treated 表示干预虚拟变量而通过引入个体/时间的固定效应,可以将 DID 表示为 TWFE 的形式: $$ \hat{\tau}^{did} = \underset{\mu, \alpha, \beta, \tau}{argmin} { \sum_{i=1}^N \sum_{t=1}^T \big(Y_{it} - (\mu + \alpha_i + \beta_t + \tau D_{it}\big)^2 } $$
合成控制法则需要先寻找最优的权重来组合样本,使数据满足平行趋势假设;其具体的建模过程,可以表述为一下形式(为例方便与 TWFE 对比分析): $$ \hat{\tau}^{sc} = \underset{\beta, \tau}{argmin} { \sum_{i=1}^N \sum_{t=1}^T \big(Y_{it} - \beta_t - \tau D_{it}\big)^2 \hat{w}^{sc}_i } $$
合成双重差分法(Synthetic Diff-in-Diff,SDID)的公式定义如下: $$ \hat{\tau}^{sdid} = \underset{\mu, \alpha, \beta, \tau}{argmin} { \sum_{i=1}^N \sum_{t=1}^T \big(Y_{it} - (\mu + \alpha_i + \beta_t + \tau D_{it})^2 \hat{w}^{sdid}_i \hat{\lambda}^{sdid}_t \big) } $$
时间权重 $\hat{\lambda}^{sdid}_t$ 的计算公式如下:
$$
\hat{\lambda}^{sdid} = \underset{\lambda}{\mathrm{argmin}} \ ||\bar{\pmb{y}}_{post, co} - (\pmb{\lambda}_{pre} \pmb{Y}_{pre, co} + \lambda_0)||^2_2 \
\text{s.t } \ \sum \lambda_t = 1 \text{ and } \ \lambda_t > 0 \ \forall \ t
$$
个体权重 $\hat{w}^{sdid}_i$ 的计算公式如下:
$$
\hat{w}^{sdid} = \underset{w}{\mathrm{argmin}} \ ||\bar{\pmb{y}}_{pre, tr} - (\pmb{Y}_{pre, co} \pmb{w}_{co} + w_0)||^2_2 + \zeta^2 T_{pre} ||\pmb{w}_{co}||^2_2\
\text{s.t } \ \sum w_i = 1 \text{ and } \ w_i > 0 \ \forall \ i
$$
SDID 的分析
2026-01-17 19:59:00
前置知识:因果推断入门
随机实验(Randomised Experiments)
ATE=E[Y|T=1] - E[Y|T=0] = E[Y_1 - Y_0] $$
常见的随机实验:
随机对照试验(RCT),评估因果效应的金标准
RCT 的基本步骤
单盲实验:受试者不知道分组信息,但是实验者知道分组信息 双盲实验:受试者不知道分组信息,实验者也不知道分组信息
RCT 的局限性:
在线对照试验 (Online Controlled Experiments),也称为 A/B 测试
A/B 测试基本步骤
RCT 与 A/B 测试不存在显著区别,也存在术语混用的情况
集群随机实验(CRT)
CRT 基本步骤
CRT 的步骤和 RCT 基本一致,只是干预的目标从个体改为集群
以一个简单的 A/B 测试为例
Python 实现代码如下
import pandas as pd
import numpy as np
from scipy import stats
# 读取数据并随机分组
data = pd.read_csv("data/online_classroom.csv")
online = data.query("format_ol==1")["falsexam"]
face_to_face = data.query("format_ol==0 & format_blended==0")["falsexam"]
confidence=0.95, h0=0
mu1, mu2 = test.mean(), control.mean()
se1, se2 = test.std() / np.sqrt(len(test)), control.std() / np.sqrt(len(control))
diff = mu1 - mu2
se_diff = np.sqrt(test.var()/len(test) + control.var()/len(control))
# 基于z统计量进行假设检验,H0为两组结果的差异为零
z_stats = (diff-h0)/se_diff
p_value = stats.norm.cdf(z_stats)
def critial(se): return -se*stats.norm.ppf((1 - confidence)/2)
# 输出不同组别的置信区间和最终的假设检验结果
print(f"Test {confidence*100}% CI: {mu1} +- {critial(se1)}")
print(f"Control {confidence*100}% CI: {mu2} +- {critial(se2)}")
print(f"Test-Control {confidence*100}% CI: {diff} +- {critial(se_diff)}")
print(f"Z Statistic {z_stats}")
print(f"P-Value {p_value}")
# 通过线性回归直接估计虚拟变量(是否线上授课)的平均因果效应
result = smf.ols('falsexam ~ format_ol', data=data).fit()
result.summary().tables[1] # 系数为负,表示线上授课对成绩存在负面影响
在实际操作过程中,可以引入更多的混淆变量(对因果变量都存在影响的变量)来修正平均因果效应 ATE 的估计;其他优化技巧可参阅回归内生性问题
对 2 家医院进行药物疗效实验
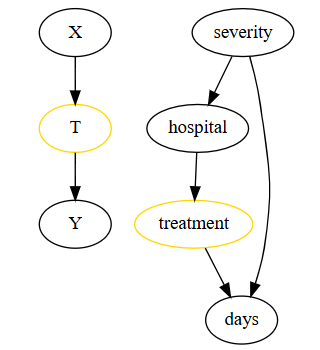
合理的混淆变量必须同时影响干预和结果;severity 满足这一点,hospital 不满足
分析催收邮件对客户还款行为的影响
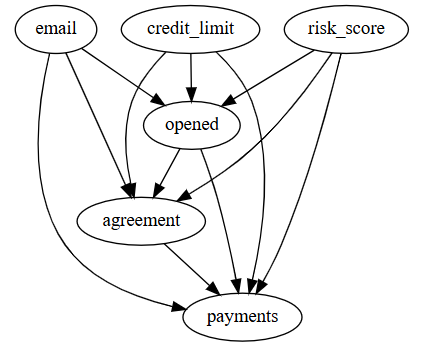
当一个特征是结果的有效预测因子,即使它不是混淆变量(同时影响干预和结果),将其纳入模型也是有好处的,能明显降低干预效果的估计方差
2026-01-14 11:05:00
去年总结: 2024年Obsidian新玩法总结
完整的 Obsidian 插件列表:Obsidian-常用插件总览
弃置不再需要或长期不维护的插件:Obsidian-已弃用插件
新增插件:
待看:
在搜索结果或知识图谱中忽略特定的文件或文件夹:
文件与链接
忽略文件选项,并进行规则添加(支持正则)其他经验技巧
Bases 工具:Dataview 查询转换为 . base 文件
IMSwitch in Math Block By XXM
在书写数学公式时自动切换输入法,以提高书写效率。当光标进入数学公式区域($ 符号之间)时,自动切换到英文输入;当光标离开公式区域时,则切换回之前的输入法。
Git Changelog By shumadrid
通过 Git 在侧边栏动态展示知识库与文件的变更历史记录,有效检测数据异常丢失。
Related Notes By Oluwasanya Awe
自动使用成熟的相似性算法分析笔记内容,以发掘库中笔记之间的连接。
MP Preview MP By Yeban
一键将 Obsidian 笔记转换为微信公众号格式且可实时预览编辑效果(自带模板较为简单,需要人工复制到公众号界面,但是支持自定义模版,可以 mark 一下)
2026-01-12 18:10:00
前置知识:因果效应评估_准实验、因果效应评估_异质性 、双重机器学习 DML
双向固定效应(Two Way Fixed Effect,TWFE)
当出现不同组的干预时间不同的情况,即错位实施(Staggered Adoption),TWFE 对干预效应的评估容易存在偏差,尤其是干预效应存在时间异质性的情况
简单来说,TWFE 会将早期干预组的干预效应看作“共同的时间趋势”,从而导致晚期干预组的效应估计被严重低估;此问题的处理将放在下文的"TWFE 改进"一章中进行讨论
案例说明:
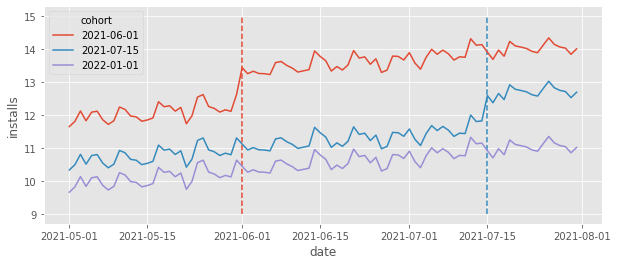
TWFE 的公式定义如下: $$ Install_{it} = \tau D_{it} + \gamma_i + \theta_t + e_{it} $$
由于虚拟变量过多,因此考虑借助双重机器学习 DML 的思想,对回归模型进行拆分: $$ \begin{align} \tilde{Installs}_{it} &= Installs_{it} - \underbrace{\frac{1}{T}\sum_{t=0}^T Installs_{it}}_\text{Time Average} - \underbrace{\frac{1}{N}\sum_{i=0}^N Installs_{it}}_\text{Unit Average} \\ \ \\ \tilde{D}_{it} &= D_{it} - \frac{1}{T}\sum_{t=0}^T D_{it} - \frac{1}{N}\sum_{i=0}^N D_{it} \end{align} $$ 最后用 $\tilde{Installs}{it} = \tau \tilde D\{it} + e_{it}$ 来快速评估干预效应
已知当干预效应存在时间异质性,TWFE 会存在严重的效应估计偏差问题
为了解决这种情况,考虑对 TWFE 的函数形式进行改进,以捕捉时间异质性: $$ Installs_{it} = \sum_{g=0}^G \sum_{t=0}^T \tau_{gt} D_{it} + \gamma_i + \theta_t + e_{it} $$
更进一步地思考,干预前的日期效应是可以忽略的,对照组的日期效应也是可以忽略的;因此可以对 TWFE 的函数形式进一步简化,来减少需要处理的参数量: $$ Installs_{it} = \sum_{G=q}^g \sum_{t=g}^T \tau_{gt} D_{it} + \gamma_i + \theta_t + e_{it} $$
2026-01-04 10:17:00
仅搜集收录了部分个人感兴趣的文章,并进行简单记录
2025-10-12 文章链接
一项针对 13,282 名癌症患者的真实世界数据显示
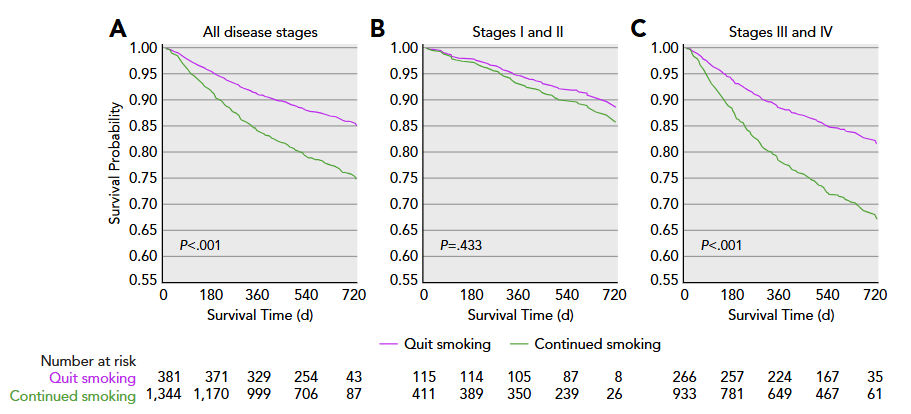
2025-10-15 文章链接
一项发表于《美国科学院院刊》(PNAS)的大规模研究,涉及英国和澳大利亚近五十万人;研究者对其中超过40万名39岁到73岁的参与者进行了多年随访,大约1%的参与者表示他们从未发生过性行为。另一项覆盖13 500名18至89岁澳大利亚居民的较小数据集也给出了类似的比例
主要结果
一种可能的解释是,认知能力较高的人往往把学习或事业放在首位,有时会牺牲亲密关系。另一种可能是,与智力相关的某些社会或人格特质——例如内向或规避风险的倾向——也可能让他们更难找到伴侣
2025-10-27 文章链接
美国心脏协会、欧洲心脏病学会和世界卫生组织(AHA/ESC/WHO)的指南一致建议:男女每周进行 150 分钟的中高强度身体活动(MVPA)
本研究分析了 80,243 名无冠心病参与者的加速度计测量的 MVPA 数据,以评估冠心病的发病率,并分析了 5,169 名冠心病患者的数据,以评估全因死亡率
主要结论:
2025-09-29 文章链接
脓毒症休克的诊断(MIMIC-IV 数据库):
mimiciv_hosp(医院模块)的 diagnoses_icd 表数据说明:
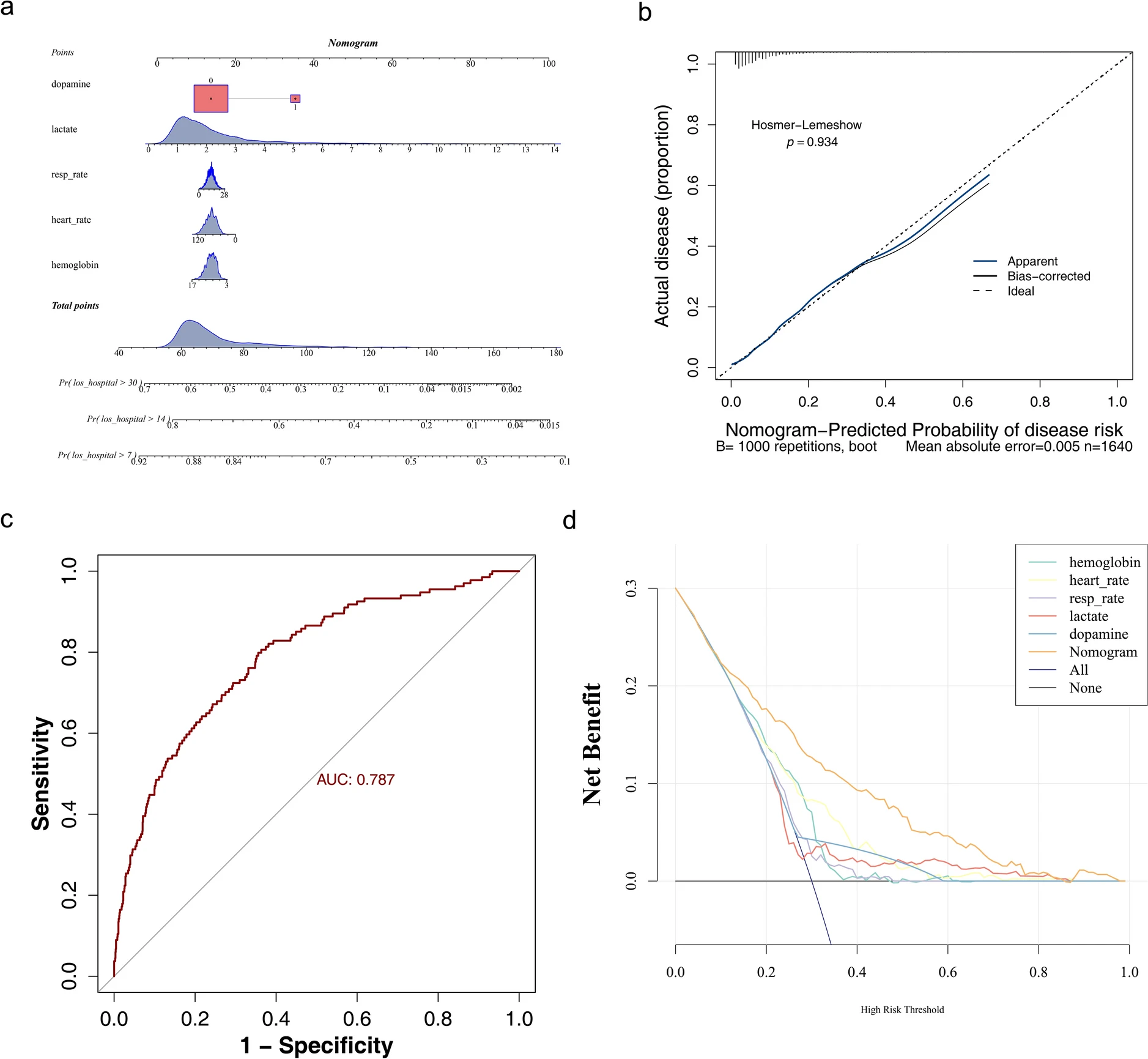
主要结论:在同时伴有低温的脓毒症休克患者与无低温的脓毒症休克患者之间,多个协变量存在显著差异,如血红蛋白、碳酸氢盐和心率;后续分析显示,血红蛋白、心率、呼吸频率、乳酸和去甲肾上腺素与低温并发症的脓毒症休克的预后相关。此外,构建的列线图显示出在有效评估低温并发症的脓毒症休克方面的良好潜力,其曲线下面积(AUC)为 0.787
2025-12-08 文章链接
目的:评估智能输血系统在术中血液管理中的长期应用效果,比较其与临床医生决策的准确性和合理性的差异
方法:回顾性分析 2017—2024 年 26 760 台手术数据,比较系统应用前后(2017—2019 vs 2020—2024)的输血预测准确率、用血合理率及临床指标变化情况。系统基于 XGBoost 算法整合患者检验参数、手术信息等数据预测红细胞输注需求。
结果:智能输血系统预测患者是否输血准确率达 80.62%,高于医生的 24.83%(P<0.001);用血合理率为83.92%,高于医生的18.02%(P<0.001)。智能输血系统应用后,三、四级手术量增加而平均申请血量下降,医生依从率>75%时输血合理率提升至 88.18%。
结论:智能输血系统显著提升输血决策精准性,减少红细胞过度使用,优化围术期输血管理,并提高血液医疗资源利用率
2023-04-03 文章链接
实验设计:
实验方法:
关键定义:
实验结论:
2025-10-27 文章链接
一、初始复苏:防止不足
脓毒性休克早期几乎所有患者存在相对低血容量(由于血管扩张)和可能的绝对低血容量(如腹膜炎、呕吐、腹泻造成的液体丢失)。如果复苏不足: 心输出量无法提升;组织缺氧持续存在;乳酸无法下降;多器官功能障碍不可逆 复苏不足的致命性,往往在数小时内表现为死亡,而液体过负荷的危害通常是可逆的中期并发症。 2. 初始剂量的思考 并非机械性执行“30 mL/kg”,而是结合病因与失液量: 腹膜炎/消化道穿孔 → 绝对缺液明显,应在 30 mL/kg 基础上适度增加 肺炎等单纯感染 → 以相对血管扩张为主,可适度下调 初始阶段不宜等待容量反应性测试,否则可能延误复苏时机。
结论:初期不怕适度多补,但千万不能少给
二、优化阶段:避免过量
随着液体输入,患者很快会进入“非反应性”状态,此时盲目继续补液只会导致水肿。动态评估至关重要:
去甲肾上腺素不仅升压,还能通过增加静脉回流、提升平均循环充盈压,起到“类液体”的作用。 对于重度低血压、舒张压极低 (<40 mmHg) 的患者,应尽早联合 NE,而不是单纯追求大剂量补液。 2. 停止补液的信号
三、稳定期:精准监测与目标调整
组织灌注:皮肤花斑消退、毛细血管再充盈时间缩短 代谢改善:乳酸下降、ScvO₂上升、二氧化碳差值改善 循环改善:心输出量增加,而非单纯血压上升 2. ICU 中的监测 热稀释法、肺动脉导管:重症患者的金标准 超声心动图:评估左室充盈压、心排出量 动脉波动指标:PPV、SVV 变化 组织氧合指标:乳酸、ScvO₂、二氧化碳差
四、清除液液阶段:防止后遗症
R(Resuscitation):复苏 O(Optimization):优化 S(Stabilization):稳定 E(Evacuation):清除 真正避免液体过负荷的关键在于“主动清除液体”。 2. 液体清除的适应证 高 CVP、肺外水增加、肺 B 线显著、腹腔压升高 影像学证据:肺淤血、全身水肿 3. 液体清除方式 利尿剂:首选 CRRT 超滤:少尿或无尿患者 白蛋白+利尿剂:低白蛋白血症合并水肿患者更有效 4. 液体清除的安全性 液体清除前必须确认“安全性”,否则极易引起循环崩溃。 可通过 PLR 或 mini-fluid challenge 等方法确认
2025-11-18 文章链接
LIBERATE-D 是一项多中心、随机、平行对照临床试验,由美国加州大学旧金山分校(UCSF)牵头。
入组标准包括:
两组方案如下:
结局指标:
主要结论: